在 Amazon SageMaker 中使用高效调优模型的 CRISPR
预测CRISPRCas9引导RNA效率的高效模型
关键要点
CRISPR技术将彻底改变基因编辑,是理解和治疗疾病的关键。通过使用预训练的基因组大语言模型LLM,可以提高gRNA效率的预测精度。本文使用了低秩适配LoRA的方法对DNABERT模型进行微调,以便在效率预测中发挥作用。此方法在预测CRISPRCas9 RNA序列的效率方面展现出了一定的优势。CRISPR成簇的规律间隔短回文重复技术有望彻底改变基因编辑技术,改变我们理解和治疗疾病的方式。该技术基于细菌中发现的自然机制,允许与单个引导RNAgRNA链相结合的蛋白质在目标基因组的特定位置进行寻找和切割。能够计算机预测gRNA的效率和特异性是基因编辑成功的核心。
转录自DNA序列的RNA是一种重要的生物序列,由核糖核苷酸A、U、G、C组成,并折叠成三维结构。得益于大语言模型LLMs的最新进展,通过对已知生物序列进行预先训练,我们可以解决多种计算生物学任务。尽管对RNA的后续任务研究相对不足,但最近的进展为此提供了可能。
在此文中,我们采用预训练的基因组LLMs来进行gRNA效率预测。我们希望通过将计算机设计的gRNA视为一句句子,来对LLM进行微调,使其能够执行类似情感分析的句子级回归任务。我们使用参数高效微调方法,以减少这一任务的参数和GPU使用。
解决方案概述
大型语言模型LLMs由于其在编码自然语言的语法和语义方面的能力,受到了广泛关注。LLMs背后的神经架构是变换器Transformer,由基于注意力的编码器解码器模块组成,可以生成其训练所用数据的内部表示编码器,并能够在同一潜在空间中生成与原始数据相似的序列解码器。鉴于其在自然语言处理中的成功,最近的研究也探索了LLMs在分子生物学信息中的应用,这些信息本质上是序列化的。
DNABERT是一个预训练的变换器模型,其使用无重叠的人类DNA序列数据。该模型的基本架构是由12个编码层组成的BERT架构。该模型的作者报告称,DNABERT能够捕捉人类基因组的良好特征表示,从而能够在后续任务中实现先进的性能,如启动子预测和剪接/结合位点识别。我们决定将该模型作为实验的基础。
尽管LLMs在市场上得到广泛应用,但微调这些模型却可能面临困难,因为需要大量的参数和计算。因此,开发了参数高效微调PEFT方法。在本文中,我们使用其中一种称为LoRA低秩适配的方法。接下来,我们将在以下部分介绍此方法。
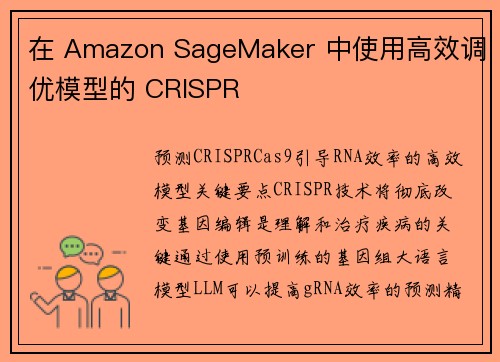
以下图示展示了Cas9 DNA靶向机制。gRNA是帮助靶向切割位点的组成部分。
本解决方案的目标是微调基本的DNABERT模型,以预测不同gRNA候选者的活性效率。因此,我们的解决方案首先收集并处理gRNA数据,之后利用Amazon SageMaker的Notebook与Hugging Face PEFT库来微调DNABERT模型,使用的RNA数据为处理后的数据。我们希望预测的标签是效率分数,该分数在实验条件下通过在细胞培养中测试实际RNA序列而计算得出。这些分数描述了能够编辑基因组与不损害未靶向DNA之间的平衡。
下图展示了所提方案的工作流程。
前提条件
要实施该解决方案,您需要访问以下内容:
一个SageMaker Notebook实例我们在mlg4dn8xlarge实例上使用单个NVIDIA T4 GPU训练了模型transformers4341peft050DNABERT 6数据集
在本文中,我们使用了研究人员在关于gRNA预测使用深度学习的论文中发布的gRNA数据集。该数据集包含针对不同gRNAs计算的效率分数。在本节中,我们描述了我们为这一任务创建训练和评估数据集的过程。
要训练模型,需要30mer gRNA序列和效率分数。kmer是从较长DNA或RNA序列中提取的一组相连的k个核苷酸。例如,如果您有DNA序列“ATCGATCG”,选择k=3,那么该序列中的kmer将是“ATC”、“TCG”、“CGA”、“GAT”和“ATG”。
效率分数
首先需要从CRISPRon论文中的补充数据1部分获取Excel文件41467202123576MOESM4ESMxlsx。在此文件中,作者发布了gRNA20mer序列和相应的totalindeleff分数。我们特别使用了名为spCas9effD10dox的工作表中的数据,并且使用totalindeleff列作为效率分数。
训练和验证数据
根据20mers及相应的crispron分数与totalindeleff分数相同,完成以下步骤以整理训练和验证数据:
 将“TRAP12K微阵列寡核苷酸”工作表中的序列转换为fasta文件。运行脚本get30mersfromfapy来自CRISPRon GitHub仓库,从步骤1获得的序列中提取所有可能的23mer和30mer。使用脚本CRISPRspecCRISPRoffpipelinepy来自CRISPRon GitHub仓库,提取步骤2中的23mer的结合能。有关如何运行该脚本的更多详细信息,请查阅CRISPRon论文作者发布的代码查看脚本CRISPRonsh。到此为止,我们得到了相应的结合能分数的23mer,以及相应的CRISPRon分数的20mer。此外,我们还有来自步骤2的30mer。使用脚本preparetraindevdatapy来自我们发布的代码创建训练和验证拆分。运行该脚本将生成两个文件:traincsv和devcsv。
将“TRAP12K微阵列寡核苷酸”工作表中的序列转换为fasta文件。运行脚本get30mersfromfapy来自CRISPRon GitHub仓库,从步骤1获得的序列中提取所有可能的23mer和30mer。使用脚本CRISPRspecCRISPRoffpipelinepy来自CRISPRon GitHub仓库,提取步骤2中的23mer的结合能。有关如何运行该脚本的更多详细信息,请查阅CRISPRon论文作者发布的代码查看脚本CRISPRonsh。到此为止,我们得到了相应的结合能分数的23mer,以及相应的CRISPRon分数的20mer。此外,我们还有来自步骤2的30mer。使用脚本preparetraindevdatapy来自我们发布的代码创建训练和验证拆分。运行该脚本将生成两个文件:traincsv和devcsv。
数据样例如下:
idrnacrisproffscorecrispronscoreseq2875p129GTCCAGCCACCGAGACCCTGTGTATGGCAC24744840998902058596491228seq2972p129AAAGGCGAAGCAGTATGTTCTAAAAGGAGG172162284931960739481132075
gRNA编码的模型架构
为了编码gRNA序列,我们使用DNABERT编码器。DNABERT在人体基因组数据上进行过预训练,因此它非常适合编码gRNA序列。DNABERT将核苷酸序列标记为重叠的kmers,每个kmer作为DNABERT模型词汇中的一个单词。gRNA序列被分解为kmers序列,然后每个kmer在输入层被kmer的嵌入替代。否则,DNABERT的架构与BERT类似。在编码gRNA后,我们使用[CLS]标记的表示作为gRNA序列的最终编码。为了预测效率分数,我们使用了一个附加的回归层。均方误差MSE损失将作为训练目标。以下是DNABertForSequenceClassification模型的代码片段:
pythonclass DNABertForSequenceClassification(BertPreTrainedModel) def init(self config) super()init(config) selfnumlabels = confignumlabels selfconfig = config
selfbert = BertModel(config) classifierdropout = ( configclassifierdropout if configclassifierdropout is not None else confighiddendropoutprob ) selfdropout = nnDropout(classifierdropout) selfclassifier = nnLinear(confighiddensize confignumlabels) # 初始化权重并进行最终处理 selfpostinit()def forward( self inputids Optional[torchTensor] = None attentionmask Optional[torchTensor] = None tokentypeids Optional[torchTensor] = None positionids Optional[torchTensor] = None headmask Optional[torchTensor] = None inputsembeds Optional[torchTensor] = None labels Optional[torchTensor] = None outputattentions Optional[bool] = None outputhiddenstates Optional[bool] = None returndict Optional[bool] = None) gt Union[Tuple[torchTensor] SequenceClassifierOutput] r labels (torchLongTensor of shape (batchsize) optional) 计算序列分类/回归损失的标签。索引应在[0 confignumlabels 1]范围内。如果confignumlabels == 1,则计算回归损失均方损失,如果confignumlabels gt 1,则计算分类损失交叉熵。 returndict = ( returndict if returndict is not None else selfconfigusereturndict ) outputs = selfbert( inputids attentionmask=attentionmask tokentypeids=tokentypeids positionids=positionids headmask=headmask inputsembeds=inputsembeds outputattentions=outputattentions outputhiddenstates=outputhiddenstates returndict=returndict ) print(bert outputs outputs) pooledoutput = outputs[1] pooledoutput = selfdropout(pooledoutput) logits = selfclassifier(pooledoutput) loss = None if labels is not None if selfconfigproblemtype is None if selfnumlabels == 1 selfconfigproblemtype = regression elif selfnumlabels gt 1 and ( labelsdtype == torchlong or labelsdtype == torchint ) selfconfigproblemtype = singlelabelclassification else selfconfigproblemtype = multilabelclassification if selfconfigproblemtype == regression lossfct = MSELoss() if selfnumlabels == 1 loss = lossfct(logitssqueeze() labelssqueeze()) else loss = lossfct(logits labels) elif selfconfigproblemtype == singlelabelclassification lossfct = CrossEntropyLoss() loss = lossfct(logitsview(1 selfnumlabels) labelsview(1)) elif selfconfigproblemtype == multilabelclassification lossfct = BCEWithLogitsLoss() loss = lossfct(logits labels) if not returndict output = (logits) outputs[2] return ((loss) output) if loss is not None else output return SequenceClassifierOutput( loss=loss logits=logits hiddenstates=outputshiddenstates attentions=outputsattentions )
微调与提示基因组LLMs
微调模型的所有参数是昂贵的,因为预训练模型体积较大。LoRA是一种创新的技术,旨在解决微调非常大型语言模型所面临的挑战。LoRA提供了一种解决方案,建议在每个变换器模块中固定预训练模型的权重,同时引入可训练层称为秩分解矩阵。这一方法大大减少了需训练的参数数量,并降低了GPU内存需求,因为大多数模型权重无需计算梯度。
因此,我们采用LoRA作为DNABERT模型的PEFT方法。LoRA在Hugging Face PEFT库中实现。当使用PEFT来训练一个模型时,可以按照如下定义低秩适配过程的超参数和包装基础变换器模型的方式:
pythonfrom peft import LoraConfig
tokenizer = AutoTokenizerfrompretrained( datatrainingargsmodelpath dolowercase=False )
DNABertForSequenceClassification是基于DNABERT架构构建的序列分类任务模型类
model = DNABertForSequenceClassificationfrompretrained( datatrainingargsmodelpath config=config )
定义LoRA配置
LORAR = 16LORAALPHA = 16LORADROPOUT = 005peftconfig = LoraConfig( r=LORAR # 低秩矩阵的维度 loraalpha=LORAALPHA # 权重矩阵的缩放因子 loradropout=LORADROPOUT # LoRA层的丢弃概率 bias=none tasktype=SEQCLS )model = getpeftmodel(model peftconfig)
评估性能
我们使用RMSE、MSE和MAE作为评估指标,并测试了秩为8和16的情况。此外,我们还实施了一种简单的微调方法,即在DNABERT嵌入后添加多个稠密层。下表总结了结果。
方法RMSEMSEMAELoRA (秩=8)119331423977014LoRA (秩=16)13039170017157一层稠密层154352382659351三层稠密层154352382419505CRISPRon117881389717134当秩为8时,我们有296450个可训练参数,大约占总量的33。此时的性能指标为:“rmse”:11933,“mse”:142397,“mae”:7014。
而当秩为16时,我们有591362个可训练参数,占总量约66。此时的性能指标为:“rmse”:13039,“mse”:170010,“mae”:7157。该设置下可能存在过拟合问题。
我们还比较了添加几个稠密层时的表现:
添加一层稠密层后,性能指标为:“rmse”:15435,“mse”:238265,“mae”:9351添加三层稠密层后,性能指标为:“rmse”:15435,“mse”:238241,“mae”:9505最后,我们与现有的CRISPRon方法进行了比较。CRISPRon是基于CNN的深度学习模型,其性能指标为:“rmse”:11788,“mse”:138971,“mae”:7134。
可以预见,LoRA的表现明显优于单纯地添加几个稠密层。尽管目前LoRA的性能稍逊于CRISPRon,但经过全面的超参数搜索,它更有可能超越CRISPRon。
白鲸加速官方在使用SageMaker Notebook时,您可以灵活保存训练期间生成的工作和数据,关闭实例并在准备继续工作时重新打开,而不会丢失任何产物。关闭实例可以避免因未使用的计算而产生的费用。我们强烈建议仅在主动使用时打开实例。
结论
在本文中,我们展示了如何使用PEFT方法在SageMaker上微调DNA语言模型。我们重点关注了预测CRISPRCas9 RNA序列效率,以期在当前基因编辑技术中产生影响。我们还提供了可以帮助您在AWS上快速启动生物学应用的代码。
欲了解更多关于医疗保健和生命科学领域的信息,请参考在Amazon EC2上运行AlphaFold v20或微调在Amazon SageMaker上微调和部署ProtBERT模型进行蛋白质分类的

